一、应用背景
毕赤酵母是现在疫苗的创新型抗体表达应用最广泛的表达系统之一。酵母在现代工业中应用非常广泛[1],与人们生活密不可分。常用于环境改造(生物修复,污染物降解)、生物制造(作物保护/食品与饲料安全/益生菌制作)、生物医药(药物发现/耐药性与代谢/疾病机制阐明)、发酵(啤酒/酱油等食品发酵)、食品与饲料配料 / 食品与饲料原料(酶制剂/食用香精/食用色素)、生物催化(手性化学中间体/生物转化),还有现代应用最前沿的异源蛋白生产(药物/激素/疫苗)[2] 见图1。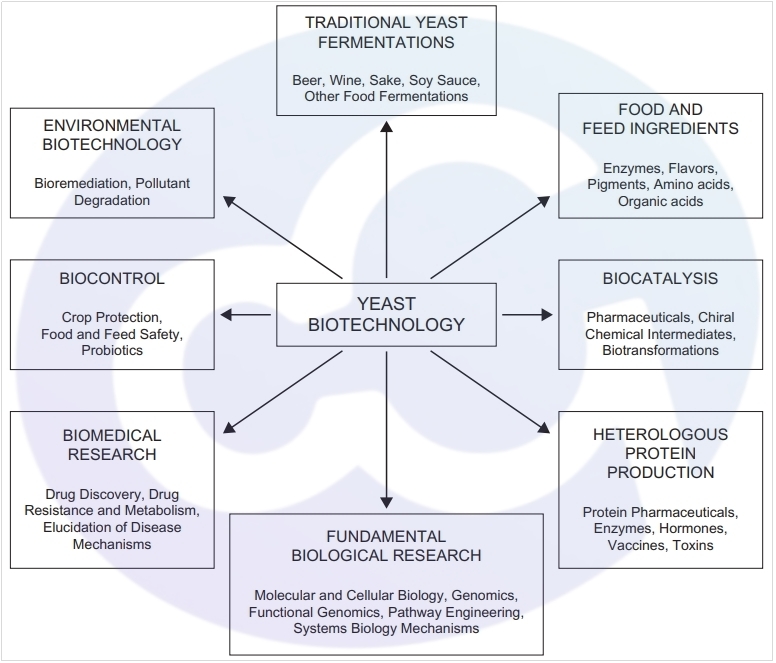
图1
经过上百年的改造和发展,毕赤酵母已经慢慢适应了多种药物的表达,但是不同的基因型菌株,对多种药物的表达量,修饰类型,所产生的效果差异较大。
毕赤酵母有着清晰的遗传背景,在蛋白折叠和转录后糖基化修饰表现优异。在寡糖修饰方面,毕赤酵母的每个侧链修饰甘露糖数量为8-14,较之酿酒酵母的50-150个糖数量短。且O-link糖基化修饰很小,也潜在避免了过度糖基化修饰。同时,由于毕赤酵母一些关键共结合因子,所以形成了独特的甲醇代谢通路,见图2。所以,部分药物表达和生产选择毕赤酵母表达系统[3,4,5,6]常见的毕赤酵母表达的药用蛋白见表1[7]: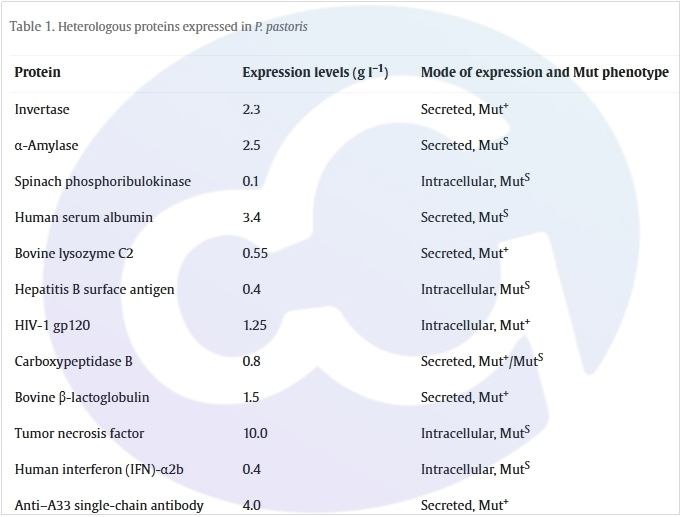
表1
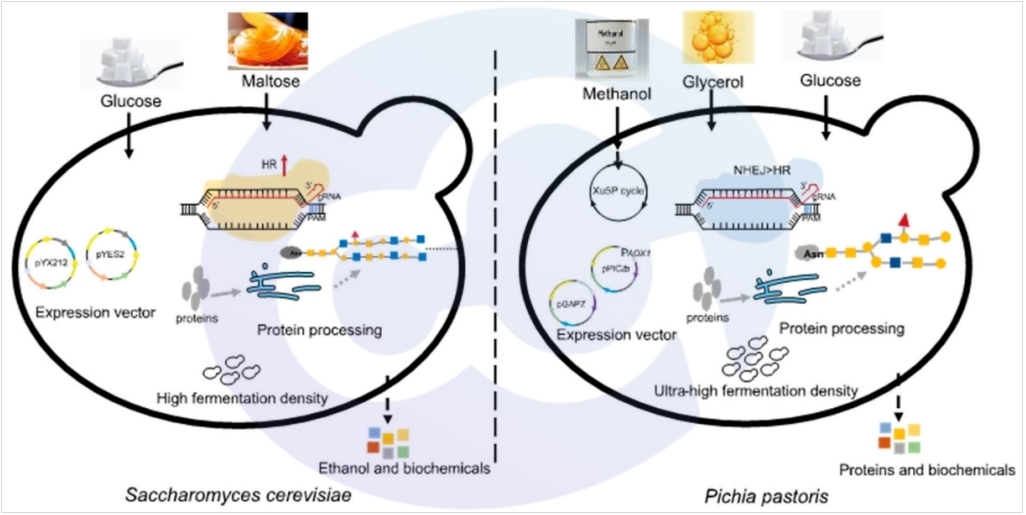
图2
二、遗传背景
所有的毕赤酵母,最初都是来源于Northern Regional Research Laboratories, (NRRL; Peoria, IL)的Y-11430野生种系。而X-33是Invitrogen的野生品系,HIS4 基因编码一种多功能酶(磷酸核糖-AMP环化水解酶、磷酸核糖-咪唑甘油磷酸脱水酶、组氨醇脱氢酶),催化组氨酸生物合成途径中的三个关键步骤。经过HIS4突变后,菌株没有了功能性的 HIS4 基因,酵母就无法自行合成组氨酸。即组氨酸影响缺陷型菌株品系,形成了GS115品系[8,9]。
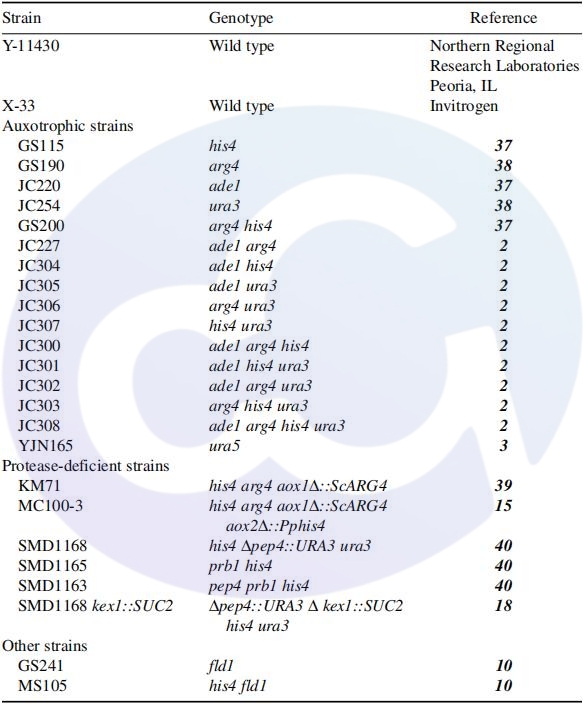
表2
现在常用的毕赤酵母有K. phaffii X33(Mut+, HIS4, AOX1, AOX2),K. phaffii GS115(Mut+, his4, AOX1, AOX2)。其中X33和GS115也有多种常见的基因型修饰:
X33, AOX1:: PAOX1-α-factor-hLALBA-TAOX1;
X33, AOX1:: PAOX1-α-factor-hLALBA-TAOX1(6 copies);
GS115, ICL1:: PICL1-LacI-Mit1AD-TAOX1;
GS115-ICL1-LM, his4:: lacO-cPAOX1-α-factor-hLALBA-TAOX1;
GS115-ICL1-LM, his4:: 5lacO-cPAOX1-P2-hLALBA-TAOX1等[10]。
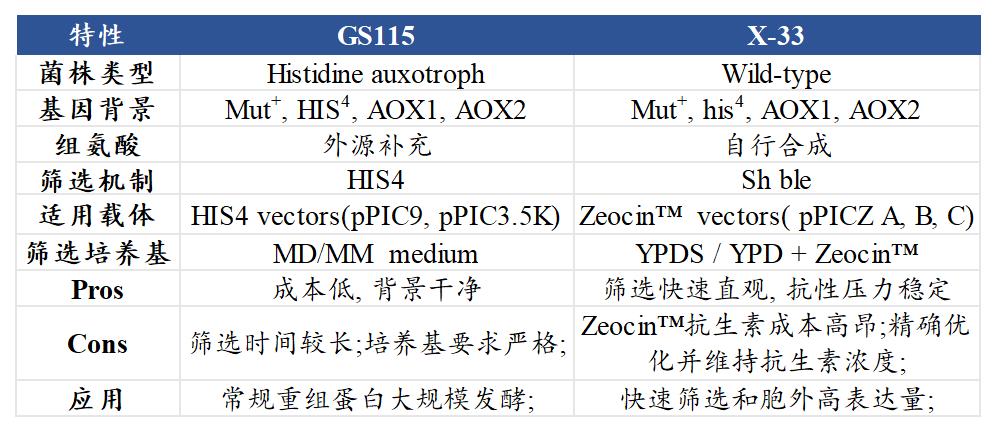
表3
三、质控数据
无论汉逊酵母、酿酒酵母,甚至是毕赤酵母,抑或是同是毕赤酵母来源的X-33和GS115品系,即便都有很近的亲缘关系,但是各自的表达谱也是存在显著的差异。GS115与X-33的遗传背景、筛选标记、营养类型、抗性和应用场景存在明显不同之处。同时,在不同药物的多样性的纯化工艺中的HCP残留,更不能同日而语。在往期的酵母之间,我们已经实验论证过其之间的交叉反应、空白本底、定量限、抗干扰等性能,详见:
《生物制药质控福音:高特异性酵母HCP ELISA方阵解决残留检测难题》
《填补市场空白!汉逊酵母 HCP ELISA,破解表达系统药物质控难题》
为了满足国内外GS115及其基础上进行基因改造后的菌株的HCP检测,我们开发了专门针对GS115毕赤酵母的HCP ELISA试剂盒(货号:PH-E0025-3A1)。通过对比X-33试剂盒,检测结果存在显著的差异。说明GS115与X-33,无论是从遗传背景,还是样本检测差异,都不能相互混用。
赛唐生物开发的毕赤酵母(GS115)HCP ELISA试剂盒,DL与QL分别在0.39ng/mL和1.56ng/mL,见图3。批内和批间CV控制在10%以内。在Tris,Sodium chloride,Urea,isopropanol,Arginine等条件下的回收率都控制在70%-130%范围,多数控制在80%-120%范围,说明本试剂盒无论是灵敏度,精密度和回收率都有显著的优势,见表4、5。另外,我们用PH-E0025-3A1,也比较了Cygnus F1015。对比终产物药物的浓度都有较高的HCP检出率,见图4。
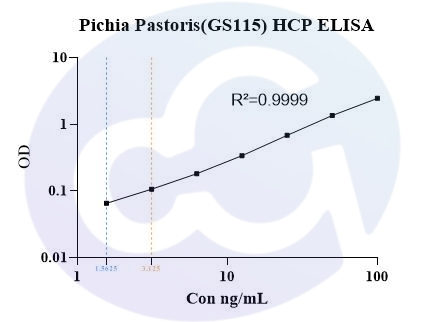
图3
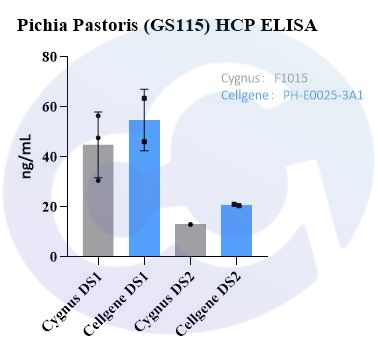
图4
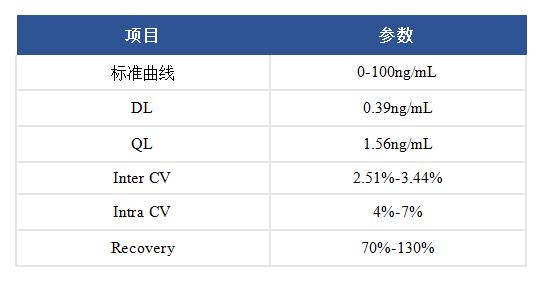
表4
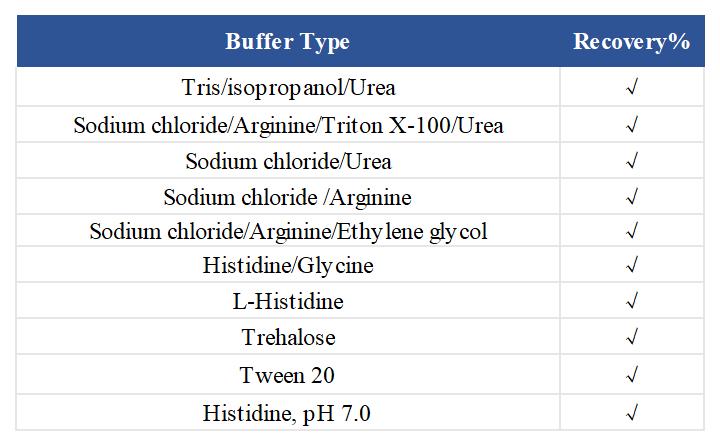
表5
参考文献:
[1]. Boud, D., & Walker, D. (1998). Promoting Reflection in Professional Courses: The Challenge of Context. Studies in Higher Education, 23, 191-206.
[2]. Cletus P. Kurtzman, et al. The Yeasts, a Taxonomic Study, Fifth Edition. Elsevier. 2011. 1:22.
[3]. Pan, Y.; Yang, J.; Wu, J.; Yang, L.; Fang, H. Current advances of Pichia pastoris as cell factories for production of recombinant proteins. Front. Microbiol. 2022, 13: 1059777.
[4]. Gao, J.; Jiang, L.; Lian, J. Development of synthetic biology tools to engineer Pichia pastoris as a chassis for the production of natural products. Synth. Syst. Biotechnol. 2021, 6, 110-119.
[5]. Grinna, L.S.; Tschopp, J.F. Size distribution and general structural features of N-linked oligosaccharides from the methylotrophic yeast, Pichia pastoris. Yeast 1989, 5, 107-115.
[6]. Zhu, T.; Zhao, T.; Bankefa, O.E.; Li, Y. Engineering unnatural methylotrophic cell factories for methanol-based biomanufacturing: Challenges and opportunities. Biotechnol. Adv. 2020, 39: 107467.
[7]. C.A. Batt. Encyclopedia of Food Microbiology (Second Edition). 2014. 42-46.
[8]. Cregg, J. M., Barringer, K. J., Hessler, A. Y., and Madden, K. R. (1985) Pichia pastoris as a host system for transformations. Mol.Cell. Biol. 5, 3376-3385.
[9]. James M. Cregg, et al. Pichia Protocols, Second Edition. Methods in Molecular Biology. 2007. 389: 13.
[10]. Xinyi Wang, et al. Ethanol-Inducible Bioproduction of Human α-Lactalbumin in Komagataella phaffii. Agricultural and Food Chemistry. 2025. 73(15):9246-9260.

VX:13764544806
WEB:www.bluegene.cc
www.cellgenebio.com
E-mail:sales@bluegene.cc
TEL:400-882-6373 / 021-61106433




